|
|
RNA
L'acido ribonucleico (RNA o ARN)
è un polimero organico, risultante dalla polimerizzazione di ribonucleotidi
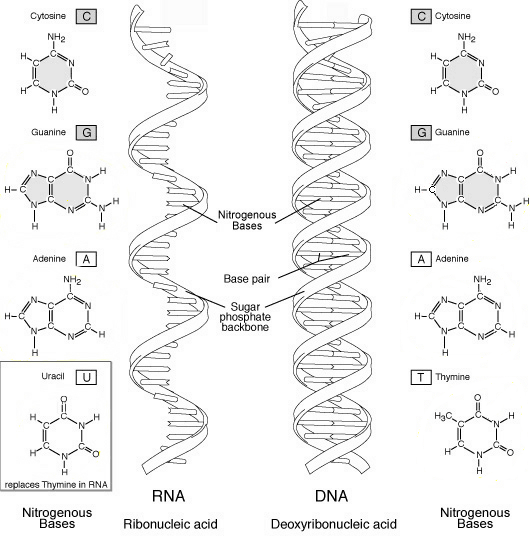
( immagine: www.accessexcellence.org
)
Chimicamente l'RNA è molto simile al DNA. Anch'esso è una catena polinucleotidica contenente quattro nucleotidi diversi. Le molecole di RNA differiscono da quelle di DNA perché:
- contengono lo zucchero ribosio (con un gruppo OH legato al carbonio 2') anziché il deossiribosio (da qui il nome);
- una delle basi, la timina, è sostituita dall'uracile (U). In questo caso è l'uracile a legarsi all'adenina, mentre la guanina si lega sempre alla
citosina;
- sono di solito a singolo filamento, anziché a filamento doppio.
Le molecole di RNA vengono sintetizzate attraverso un processo, conosciuto come trascrizione del DNA, dove un filamento di DNA viene ricopiato nel corrispondente filamento di RNA
Vi sono tre tipi di RNA comuni a tutti gli organismi cellulari:
- mRNA (messaggero) che contiene l'informazione per la sintesi delle proteine;
rRNA (RNA ribosomale), che entra nella struttura dei ribosomi;
tRNA (RNA transfer) necessario per la traduzione nei ribosomi.
Negli eucarioti abbiamo anche:
hnRNA (RNA eterogeneo nucleare) che deve subire una maturazione per divenire mRNA;
snRNA (piccolo RNA nucleare) necessario per la maturazione dell'HnRna.
La sintesi dell'RNA è molto simile a quella del DNA. La RNA polimerasi non richiede però un innesco. La trascrizione può iniziare solo presso una sequenza detta promotore e termina in presenza di altre sequenze particolari.
È stata avanzata l'ipotesi che l'RNA abbia assunto un ruolo chiave negli organismi primitivi prima del DNA. A favore di tale ipotesi c'è la capacità catalitica di alcune molecole di RNA (ribozimi).
Sull' mRNA viene trascritta l'informazione genetica che poi verra utilizzata per svariati usi.
Sintesi dell'RNA
Questo processo è molto diverso tra procarioti ed eucarioti. Negli eucarioti la sintesi avviene nel nucleo attraverso tre diverse molecole dette RNA polimerasi (nei procarioti ce n'è solo una). Sono parzialmente diverse tra loro, infatti hanno alcune subunità in comune e altre uniche per la loro specie. Ciò è anche dovuto al fatto che esse producono tre tipi diversi di RNA.
- La RNA polimerasi I produce gli rRNA 5,8 S, 18 S, 28 S nel nucleolo
- La RNA polimerasi II produce mRNA e piccoli RNA stabili che servono a formare gli snRNP nel nucleo
- La RNA polimerasi III produce piccoli RNA stabili, tRNA e l'RNA 5 S sempre nel nucleo.
- La RNA polimerasi più conosciuta è la RNA polimerasi II perché produce l'mRNA, che servirà poi ai ribosomi per sintetizzare le proteine.
L'rRNA 18 S più 35 proteine compresse andrà a formare la subunità ribosomiale minore. Gli rRNA 5 S, 5,8 S e 28 S più 50
proteine andranno a formare la subunità ribosomiale maggiore.
Quali sono le anomalie?
Durante il corso degli anni i ricercatori si sono accorti che gran parte dell'RNA sintetizzato dalle polimerasi veniva scartato e solo una piccola parte veniva inviata sotto forma di mRNA per la sintesi proteica. Infatti per una proteina media di circa 400 a.a. (quindi 1200 nucleotidi) venivano sintetizzati anche più del doppio dei nucleotidi realmente necessari.
Ciò è dovuto al fatto che nel DNA esistono delle sequenze non più codificanti, che servivano alla cellula quando non era ancora specializzata. Queste sequenze vengono comunque trascritte dalla polimerasi e vengono dette introni, quelle che invece vengono copiate e codificanti esoni. Questo fatto implica che prima della traduzione, esse andranno tagliate; in un processo denominato Splicing
Il processamento
Prima dell'eliminazione delle sequenze introniche l'RNA subisce delle ulteriori modificazioni. La prima di queste avviene quando la polimerasi ha trascritto circa i primi 30 nucleotidi. In posizione 5' viene aggiunto un cappuccio di m7G (7-metil guanosina) il cui scopo è di far si che l'RNA non vada contro degradazione e inoltre giocherà un ruolo molto importante durante la traduzione.
Una seconda modificazione consiste in un taglio di una sequenza specifica che avviene circa 10-30 nucleotidi più a monte della porzione terminale 3', esattamente dove si trova la sequenza AAUAAA. Dopodiché, dopo la porzione tagliata, un enzima chiamato poli-A-polimerasi, crea una sequenza di poli-A di circa 200 nucleotidi di adenina.
Splicing
Ora abbiamo un filamento di RNA che viene detto hnRNA (heterogenic nuclear RNA). Il fenomeno di splicing, come detto in precedenza, consiste nell'eliminazione delle sequenze introniche per portare l'hnRNA a mRNA pronto alla traduzione.
L'hnRNA dopo essere stato creato non si trova sparso nel nucleo ma viene complessato in particolari strutture simili ai nucleosomi, create mediante proteine hnRNP (che in questo caso sostituiscono gli istoni). Queste strutture sono più grandi e avvolgono 500 nucleotidi per ogni snRNP. L'intera struttura complessata è chiamata spliceosoma.
Lo splicing vero e proprio avviene tramite delle molecole di snRNP che sono strutture molto simili ai ribosomi a cui sono attaccate piccole molecole di RNA e che entrano a far parte di molti importanti meccanismi. Esse vengono catalogate come snRNP U1, U2, U3,…
Gli introni non hanno alcuna funzione ma possiedono due piccole sequenze, una a monte e una a valle che rivestono due ruoli principali. In 3' troviamo AG e in 5' troviamo GU.
Una snRNP U1 si lega a GU mentre una snRNP U2 si lega ad una A più a valle. A questo punto entra un complesso di snRNP U4/U6 + U5. La snRNP U4 si stacca da U6 che a sua volta si sostituisce ad U1. Ora snRNP U5 attacca la porzione AG in 3' tagliandola. A questo punto la porzione a monte in 5' si lega alla A a cui è attaccata la snRNP U2 creando un legame AGU. Si forma quindi una particolare struttura a lazzo, a cui sono legate le snRNP, che andrà degradato nel nucleo.
A questo punto sono stati eliminati tutti gli introni e il filamento di mRNA è pronto per essere esportato e tradotto.
RNA tranfer (tRNA)
L'RNA transfer è costituito da un filamento di RNA di circa 80 basi azotate ripiegato a formare quattro lobi, che ricordano la caratteristica forma di un quadrifoglio. Un lobo riconosce tramite un anticodone la tripletta sull'RNA messaggero corrispondente all'amminoacido trasportato dal transfer; un altro riconosce l'enzima che attacca l'amminoacido al transfer; un terzo è il sito di riconoscimento del ribosoma. Trasferisce ai ribosomi i vari amminoacidi, che uniti formano fra loro un legame peptidico per poter formare le proteine.
RNA messaggero
L'mRNA si occupa di trasportare le informazioni codificate nel DNA al citoplasma. L'mRNA si presenta sotto forma di filamento sul quale sono presenti triplette di nucleotidi (detti codoni). Il processo di formazione di un nuovo filamento è detto trascrizione. La sequenza base del filamento di mRNA è complementare a quella del filamento del DNA dal quale è stato copiato, e non identica. Quando l'mRNA ha trasmesso l'informazione si scompone nei nucleotidi che lo componevano.
RNA ribosomiale
Nel citoplasma, forma con le proteine particolari organuli, i ribosomi, sui quali avviene la sintesi proteica. Traduzione dell'RNA Il meccanismo utilizzato per la conversione dell'informazione da sequenze di nucleotidi in sequenze di amminoacidi è conosciuto come codice genetico; il processo di trasferimento di informazione dagli mRNA alle proteine costituisce la cosiddetta traduzione dell' informazione genetica. Il codice genetico consiste nell'esistenza di una corrispondenza tra unità di messaggio, costituite a sequenze di tre nucleotidi (triplette o codoni) contenute negli mRNA, e uno dei venti amminoacidi che costituiscono le proteine
Sito web consigliato:
WIKIPEDIA,
l'enciclopedia libera...

Questo articolo è protetto dalle Leggi Internazionali di Proprietà.
E' PROIBITA la sua riproduzione totale o parziale, all'interno di qualsiasi mezzo di comunicazione
(cartaceo, elettronico, ecc.) senza l'autorizzazione scritta dell'autore.
|
